随着环状RNA领域的研究越来越多,更多的环状RNA功能被发掘,例如:作为RNA适配体、miRNA和蛋白质的分子海绵、反义RNA、调控细胞内天然免疫反应、蛋白质翻译载体及疾病相关分子标志物等。这些功能也使得环状RNA在疾病的诊断和治疗方向都有广阔的应用前景。
当前医药企业已经布局的重要管线中涉及最多、推进最快的方向是将环状RNA用于目标蛋白的表达。在该方向上,企业通常会重点关注以下几个方面的问题:翻译元件的筛选、环化技术的优化,以及递送技术的探究。
其中,环状RNA与线性RNA最大的区别在于需要增加将线性RNA环化成环的步骤,因此体外环化是环状RNA领域中面临的独特技术难题,也是环状RNA是否真的能够产业化的重要一环。针对体外环化技术,不同的研究主体会开发和使用不同的策略以解决RNA环化中存在的问题。
当前的体外环化方法共有三种:化学方法,连接酶方法、以及核酶方法。
其中,化学方法主要通过缩合剂催化实现。但是,由于这种催化反应没有特异性,生成产物的稳定性差,且容易发生分子间反应产生多聚体,大部分研发不倾向于使用该技术。连接酶法通常借助T4 DNA或RNA连接酶,其能够解决化学法中反应没有特异性的问题,但是同样容易发生分子间反应,且其环化效率也会受RNA的长度和二级结构的影响,仅适用于环化最多约300个核苷酸的序列,另外需要引入一个DNA互补链或者RNA互补链作为夹板序列(DNA splint or RNA splint),将RNA分子的末端拉近,这极大增加了工艺难度。因此,这种方法从操作复杂程度和安全控制来看,通常用于实验室级别的研究,而不适用于药物生产。
基于化学方法和连接酶法所存在的问题,如何提高环状RNA的精准环化效率,实现较长插入序列,并顺利实现分子内的连接是在开发环化技术中亟待解决的难题。
当前研究最多的环化策略就是基于自剪接内含子的核酶法,这一策略利用了内含子核酶自催化剪接反应,基于不同种类内含子来构建PIE系统实现环化。
PIE(permuted intron-exon),也称置换的内含子-外显子元件,顾名思义,是指将天然的内含子、外显子剪切,置换位置并形成新的内含子-外显子构建体,在构建体中可以插入目标序列,转录后通过体外反向剪接合成环状RNA的系统。这一系统中,内含子片段会在自我剪接时凋落,两侧的外显子片段实现相连,完成RNA的体外环化。
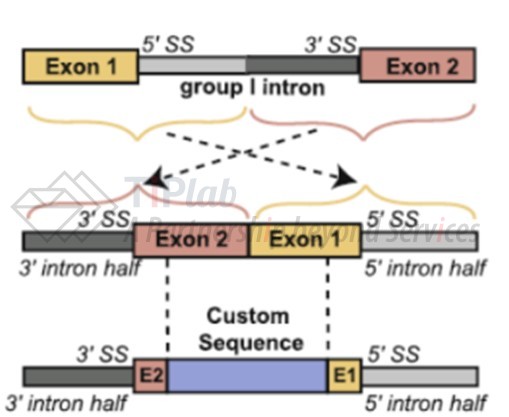
不同种类的内含子会在连接时具有不同的机理,从而在连接的产物、连接的温度以及连接的效率上有所不同。目前最常用于环化RNA的核酶包括 Ⅰ 型内含子(Group I intron)和 Ⅱ 型内含子(Group II intron)。
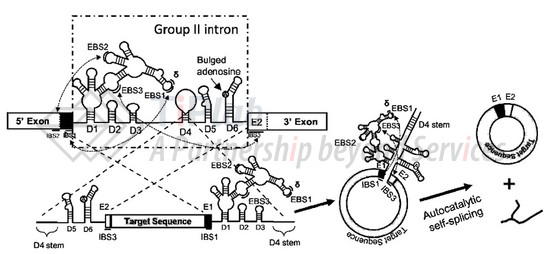
其中, Ⅰ 型内含子需要一个游离的G-OH供能,攻击内含子的5’端剪接位点,在外显子的末端会产生3’-OH,3’-OH可以去攻击内含子5’端的剪接位点,使得两个外显子相连,内含子被剪切下来。而 Ⅱ 型内含子不需要G-OH供能,内含子中特定的腺苷可以攻击5’端剪接位点,形成一个套索,5’端外显子的3’-OH攻击3’剪接位点,从而将两个外显子结合。
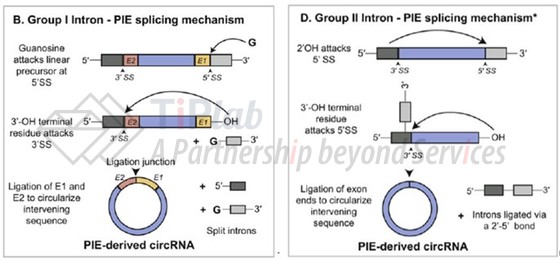
基于Group I intron 的PIE系统的构建是当前RNA体外环化工艺中使用最多的技术。1992年,M. Puttaraju和Michael D. Been基于鱼腥藻 Ⅰ 型内含子,发明了 Ⅰ 型内含子-外显子置换系统(Group I intron PIE)实现核酶催化的体外自主剪切。该技术需要外源的GTP供能来攻击E1和5’ intron的位点使其断开,产生的来自E1(Exon 1)的羟基会继续与E2(Exon 2)反应,实现E1和E2的连接。这种方法增加了环化产物的均一性,提高了环化效率。但最终的产物中,会残留E1和E2的序列,该序列通常是来自于噬菌体或细菌等原核生物的序列,可以保证环化的发生。但是这些序列在用于体内时,可能引发较大的免疫原性,也可能影响表达蛋白的活性;且该方法也会受到RNA长度和结构的限制,可环化长度只有300-500bp,使得其产业化应用受到很大阻碍。
为解决上述问题,研究人员设计了不同的改造策略来提高环化效率。例如,设计辅助序列、替换新的内含子、筛选成环位点等。
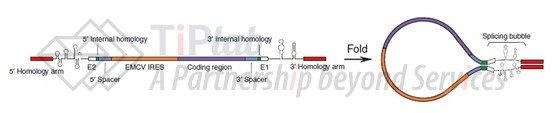
Daniel Anderson和Alexander Wesselhoeft于2018年报道了基于Group Ⅰ intron PIE的改进方案,该方案在前体RNA的5’端和3’端添加同源臂(homology arms)的设计,使剪接位点在空间上靠近,增加了剪接效率的同时将环化的RNA长度扩展到了5kb。同时,考虑到3’ intron的剪接位点离IRES(翻译起始元件)较近,且两个序列都是高度结构化,可能在空间上会互相影响,又设计添加了间隔序列(spacer),用于保留内含子的二级结构。
环码生物于2022年发表文章报道了新的环化策略,该策略使用Ⅱ 型内含子,即Group Ⅱ intron PIE技术 ,通过设计人工RNA环化核酶(cRNAzyme)用来更好地模拟真核生物体内的RNA剪接过程得到环形目标序列。此过程不需要额外的GTP供能,不仅可以仅保留外显子结合EBS序列识别的内含子结合IBS序列,极大地缩短了E1和E2的长度,还可以进一步将编码区的一部分序列视为IBS序列,对内含子的EBS序列进行相应突变和改造,在保证环化效率的同时,降低环化过程中可能引入的免疫原性增加的问题。并且,由于反应条件比较温和,该技术可以实现体外转录(IVT)共环化,极大的简化了工艺。
针对核酶法引入的外源序列残留带来的免疫原性的问题,还有一些其他的解决方案。2022年6月,科锐迈德公开了一种新的用于RNA环化的Clean-PIE系统,可以通过筛选编码区或IRES区中可以作为剪接位点的合适的位置,进行改造设计,这样就无需引入外源外显子序列E1和E2实现无外源序列残留,同时提高环化效率。
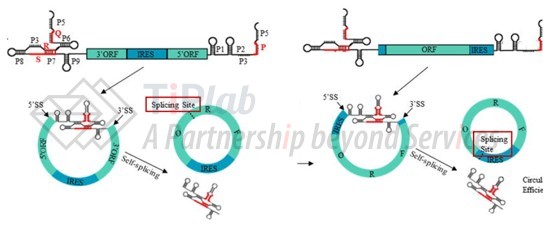
综上所述,现有的环化技术通常会考虑从以下几个方面进行优化:1)设计添加辅助序列,帮助RNA的5’端和3’端连接,在扩大目标序列长度的同时避免可能产生的分子间连接,提高连接的准确性;2)改进已有核酶的结构/序列设计,使其能够更高效地进行自剪接,尽量避免二级结构带来的对环化效率的影响,以及过多的外源序列残留带来的免疫原性的问题;3)筛选新的核酶,能够在保持较高的连接效率的同时,降低工艺难度。
当前环化技术的开发还处于非常早期的阶段,本文中我们介绍的大部分环化技术优化方案也是近两年才研发出来并投入使用的。针对环状RNA环化步骤遇到的各种问题,各研究主体还在不断地创造性地提出一些解决方案,并且也都积极地进行了相应的专利布局。下一篇文章我们将重点分析上述代表性企业针对环化技术进行了哪些专利保护。
* 以上文字仅为促进讨论与交流,不构成法律意见或咨询建议。
