在序中我们已经提到,DNA碱基编辑器技术主要衍生于CRISPR/Cas系统,这里我们先介绍CRISPR/Cas系统。
自2013年首次报道CRISPR/Cas9系统之后,科学家们又开发了多种CRISPR/Cas系统。我们以CRISPR/Cas9系统为例,其包括Cas9和引导RNA序列(sgRNA)两部分,经“定位解旋、R-Loop环的形成、切割和修复”四步实现目标基因的编辑。
具体而言,首先通过Cas9对PAM域的识别作用将Cas9/sgRNA复合物定位至目标区域,并开始促进目标区域双链DNA的解旋,解旋的双链DNA在sgRNA的参与下形成R-Loop环,随后Cas9切割开环后的两条DNA链,最后断裂的双链DNA通过非同源性末端连接(Non-Homologous End-Joining,NHEJ)或同源介导修复(Homology-Directed Repair,HDR)实现基因的编辑。
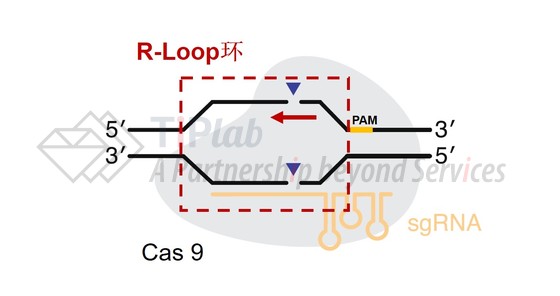
在CRISPR/Cas系统的两种修复途径中,NHEJ方式占主导,其具有一定的随机性,通常会引起一个或多个碱基的插入或缺失。而HDR修复则可以借助人工模版来实现定向编辑。
对于人类点突变遗传病来说,通常需要将错误突变的碱基纠正为正确的碱基,这就更依赖于HDR修复方式。不过机体内HDR修复的效率一般仅为0.1~5%,如何提高修复效率成为了CRISPR/Cas系统在单点突变“纠错”治疗时需要克服的技术难点。
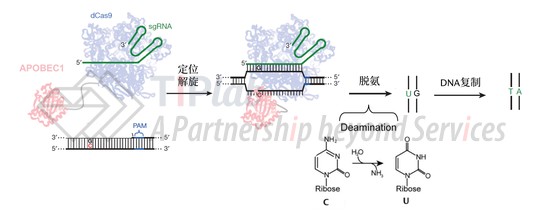
2014年,David Liu开发了第一代碱基编辑器(BE1),能够实现C→T的定向转化,其在体外蛋白水平的实验中达到了约44%的碱基编辑效率。
BE1首先依靠dCas9(一种DNA切割活性完全失活的Cas9)的定位解旋功能,在目标DNA区域结合并形成“R-Loop”环,随后dCas9上融合的胞嘧啶脱氨酶(APOBEC1)对胞嘧啶(C)进行水解脱氨形成尿嘧啶(U)。在DNA复制时,U会被读作胸腺嘧啶(T),并偏向于和腺嘌呤(A)实现碱基互补配对,从而实现C•G到T•A的定向转化(如图2所示)。
不难看出,碱基编辑器的基本结构其实是在CRISPR/Cas系统基础上引入了碱基转变酶,并且将Cas酶替换成失活的核酸酶(即,不具有双链切割活性的核酸酶)。
其中,碱基转变酶的功能就是将错误突变的核苷酸选择性转变成“中间体核苷酸”(如:BE1中的C被APOBEC1水解脱氨转变成U)。而失活的核酸酶则是保证不会产生DNA的双链断裂,此时碱基编辑器的修复方式主要依靠机体DNA复制时,“中间体核苷酸”碱基互补配对的偏向性 (如:BE1中的U在DNA复制时,会被读作T,偏向于和A互补配对)。
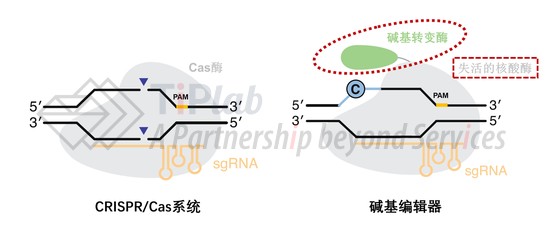
如果将CRISPR/Cas系统比作可以在基因上精准剪切的剪刀,那么碱基编辑器就是一支可以对碱基进行定点涂改修正的铅笔。通过对碱基编辑器特有的两个基本元件(碱基转变酶和失活的核酸酶)进行不同选择,可以设计出多种多样的碱基编辑器。
首先,对于碱基转变酶这一基本元件而言,顾名思义就是能够使碱基类型发生改变的酶。所以理论上,除了C→T的定向转化外,依赖于不同的碱基转变酶和碱基转变机制设计的碱基编辑器均可以实现不同类型碱基之间的相互转变。
比如,David Liu在2017年开发的腺嘌呤碱基编辑器(ABE)是选择将腺嘌呤脱氨酶作为碱基转变酶,从而实现A→G的定向转化。
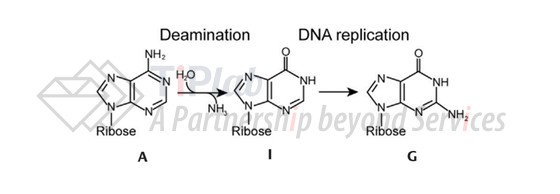
腺嘌呤脱氨酶可以将A水解脱氨转化成次黄嘌呤(I)中间体,随后在DNA复制时,I会被读作G(鸟嘌呤),与C进行碱基互补配对,就可以实现A•T到G•C的定向转化。
为了获得具有上述功能的腺嘌呤脱氨酶,David Liu最初尝试使用自然界中存在的RNA腺嘌呤脱氨酶(如:TadA),但将其应用到DNA碱基编辑器时,并没有产生预料之中的A→G转化。为此,David Liu开发了一种演化平台,对tRNA腺嘌呤脱氨酶(TadA)进行了多轮突变演化,最终筛选到了能够对DNA上的腺嘌呤进行脱氨的突变体。
此外,目前还出现了许多其它类型的碱基编辑器,包括David Liu发明的CGBE,选择尿嘧啶DNA糖苷酶(UNG)作为碱基转变酶,能够最终将胞嘧啶(C)定向转化成鸟嘌呤(G);J. Keith Joung发明的双碱基编辑器SPACE(Synchronous Programmable Adenine and Cytosine Editor),同时引入胞嘧啶脱氨酶和腺嘌呤脱氨酶作为碱基转变酶,可以同时实现C→T和A→G的定向转化。我们可以预期未来将会有更多的碱基转变酶被发现,设计出的碱基编辑器种类将更加丰富。
回到碱基编辑器的基本结构,另一个基本元件是失活的核酸酶,它的主要功能是能对双链DNA进行定位解旋,同时不产生DNA双链的断裂。那么,我们可以合理试想,对于不产生双链断裂的Cas酶,除了dCas酶外,选择只切割一条链的nCas酶也能够实现相关功能。
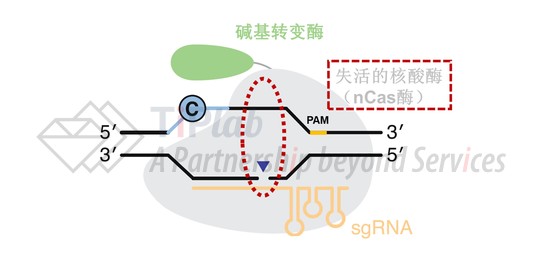
比如2017年,David Liu通过将dCas9替换成了含D10A突变的nCas9设计出了另一种碱基编辑器,并将碱基编辑器的编辑效率进一步提高到2-6倍。
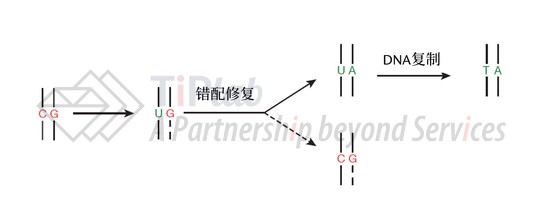
因为DNA复制时选择编辑链(即,碱基转变酶作用的DNA链)还是非编辑链作为模板进行复制均存在50%的概率,所以理论上使用dCas9的碱基编辑器最高修复效率难以达到50%。但nCas9(D10A)可以切割非编辑链也就是图6中含G的DNA链,机体会认为断裂的非编辑链是新合成的链,会以编辑链(含U的DNA链)为模板,通过错配修复(MMR)途径将G修复成A。
除了从Cas酶失活的角度进行选择外,随着不同类别和不同来源的Cas酶的出现,理论上能够定位解旋DNA的Cas酶在失活后都能够应用于碱基编辑器。
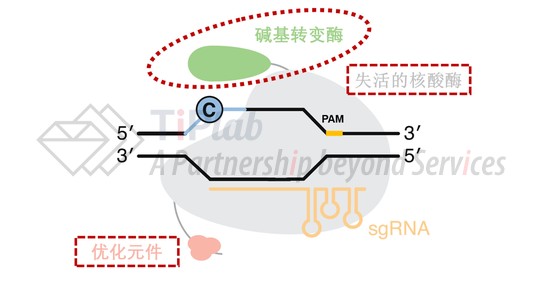
让我们再次回到碱基编辑器的结构,除了碱基转变酶和失活的核酸酶这两个基本元件外,部分碱基编辑器还存在优化元件,以提高碱基编辑器的性能。比如,添加尿嘧啶糖苷酶抑制剂(UGI)可以提高编辑效率。
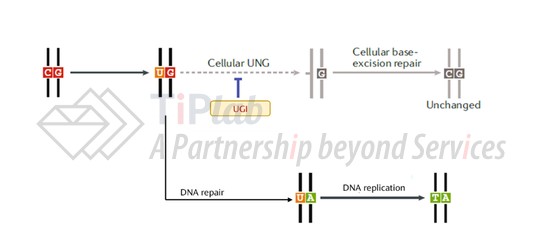
虽然前文提到BE1在体外蛋白水平已能够实现约44%的编辑效率,但在细胞内的编辑效率却只有0.8%-7.7%。究其原因其实是细胞内存在一种尿嘧啶DNA糖苷酶(UNG),当胞嘧啶脱氨酶将C水解脱氨成U后会被UNG切除,形成无碱基核苷酸位点,随后机体会通过碱基切除修复路径将无碱基位点重新修复回C,造成“竹篮打水一场空”的局面。
2016年,David Liu在碱基编辑器基本结构的基础上进一步引入了能够抑制UNG活性的UGI,在UGI的保驾护航之下,碱基编辑器的体内C→T编辑效率被成功提高至3倍
结合DNA碱基编辑器的技术原理和发展历程,我们认为其专利风险主要来源于相应的CRISPR/Cas系统、碱基编辑器基本结构以及优化元件三个角度。
碱基编辑器首先需要考虑的是来自于CRISPR/Cas系统的专利风险,因为CRISPR/Cas系统的专利权利要求通常不会限定Cas酶是否具有切割活性,换句话说,这些专利大概率会涵盖不具有切割活性Cas酶,比如dCas。特别是CRISPR/Cas9系统,以诺贝尔奖得主Jennifer和Emmanuel为代表的CVC团队和以张锋为代表的Broad团队都拥有保护范围相当宽泛的专利,在双方专利之争尚未尘埃落定前,就连Beam也为之头疼。除此之外,CRISPR/Cas领域的创新仍处于持续高涨状态,涉及多种角度优化创新的专利风险也不断增加。比如,对于不同类型和来源的Cas酶都可能存在专利保护。
其次,基本结构的专利风险包括两个基本元件单独的专利风险及其组合的专利风险。不同基本元件的组合可以形成各种碱基编辑器,相应的专利风险也不尽相同,比如David Liu保护第一代碱基编辑器基本结构的专利申请,该专利在部分国家和地区授权的权利要求仅限定为含有失活的Cas9和脱氨酶,并没有对Cas9和脱氨酶的结构做出任何限定,我们认为保护范围相当宽泛。另外,不同的碱基转变酶和失活的核酸酶还可能存在相应专利申请单独进行保护,这部分风险也不容忽视。比如,为ABE设计腺嘌呤脱氨酶时,需要考虑可能来自于David Liu单独保护其腺嘌呤脱氨酶突变体的专利风险。
此外,优化元件的专利风险也需要单独考虑,选择不同的优化元件应当考虑相对应的专利风险。比如,增加UGI作为提高编辑效率的其中一种优化方式,就需关注包括David Liu等人的相关专利申请。
可以看到,CRISPR/Cas系统、碱基编辑器基本结构以及碱基编辑器优化元件相关的专利风险,在一起构成了复杂的专利壁垒。不同碱基编辑器的Cas酶、碱基转变酶以及优化元件等均存在差异,其对应的风险也有明显区别,因此,对于特定的碱基编辑器需要具体分析。下一篇文章我们将尝试讨论解决这些风险的基本思路。
* 以上文字仅为促进讨论与交流,不构成法律意见或咨询建议。
